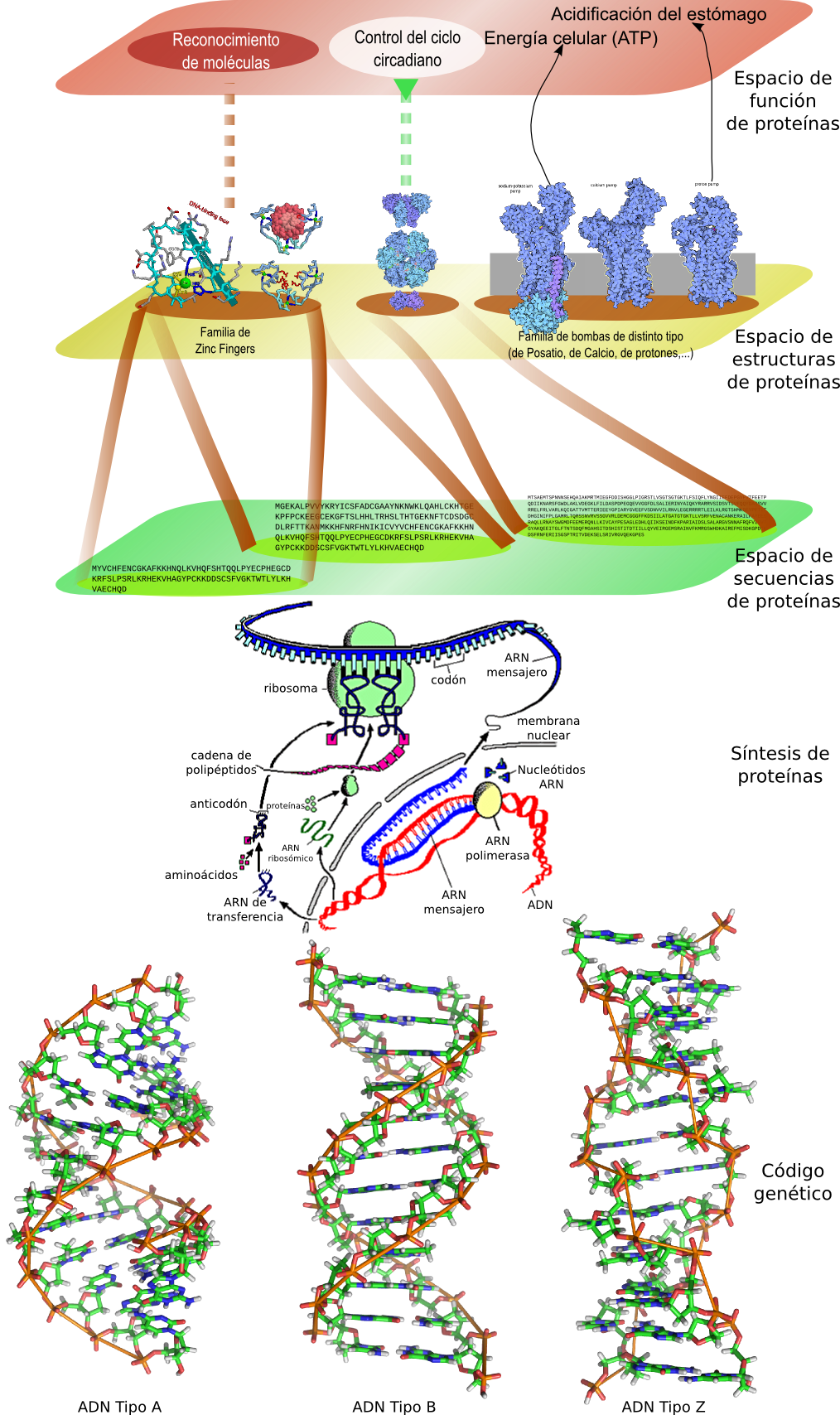
(Para los que sois nuevos en este blog, ésta es la cuarta entrega de una serie de entradas basadas en el material aportado por David, que es un artículo en Nature Reviews Genetics del año 2000 de Joel B. Hagen, titulado “The origins of bioinformatics”.)
Tras las 3 entregas anteriores, donde hemos visto cómo se descubrían y establecían algunos de los paradigmas sobre los que se sostiene la biología molecular moderna, todavía os estaréis preguntando “Éste es un blog de Bioinformática. ¿Y cuando salen los ordenadores?”. Pues ha llegado el momento de escribir sobre cómo se empezó a introducir el uso de los ordenadores, y cómo empezó a nacer la biología computacional.
A finales de los años 50 el mundo académico (en Estados Unidos, se entiende) empezó a tener acceso no restringido a los ordenadores, que hasta ese momento sólo habían tenido un uso militar. De todos los descubrimientos de esa época en el área de las ciencias de la vida, sólo se puede decir que los ordenadores tuvieron un papel determinante en la resolución de la estructura tridimensional de la mioglobina en 1957 (mediante la técnica de cristalografía de rayos X). Estos ordenadores estaban basados en válvulas de vacío, y se programaban a bajo nivel (no se podían reutilizar los programas), pero ayudaron a comenzar el desarrollo de técnicas computacionales y de desarrollo de software en el mundo académico.
Ya a principios de la década de 1960 había un acceso casi generalizado en el mundo académico a los ordenadores. Algunas universidades empezaron a comprar ordenadores de segunda generación, basados en transistores, que además empezaban a llevar lenguajes de alto nivel. El primer lenguaje en alto nivel disponible fue FORTRAN, que fue desarrollado inicialmente por IBM para sus máquinas, lo cuál permitió que se pudiera empezar a reutilizar los programas desarrollados en otros ordenadores con arquitecturas diferentes.
Y aquí entra en juego Margaret Oakley Dayhoff (sitio oficial http://www.dayhoff.cc/), una de las pioneras de la Bioinformática. Ella comenzó con una preparación en química cuantitativa y matemáticas, y alrededor de los años 60 empezó a estar interesada en el mundo de las proteínas y la evolución molecular. Tuvo un buen punto de partida para sus intereses científicos, al ser en aquella época directora asociada de la recién creada National Biomedical Research Foundation, una fundación interesada en la promoción y aplicación de las técnicas computacionales para ayudar en la investigación médica. Margaret Dayhoff empezó a explorar las distintas técnicas matemáticas para analizar los ya crecientes datos de secuencias de aminoácidos.
 A lo largo de sus investigaciones recibió fondos de NIH (National Institutes of Health), NSF (National Science Foundation), NASA (National Aeronautics and Space Administration) y la corporación IBM. Estas investigaciones se movían en varios frentes, de los cuáles el primero fue la escritura de una serie de programas en FORTRAN para determinar la secuencias de aminoácidos de las moléculas de proteínas. Estos programas tomaban fragmentos de péptidos obtenidos de la digestión parcial de una proteína, y calculaban todas las posibles secuencias compatibles con los ensamblajes de esos péptidos. Estos programas servían para resolver el mismo puzzle que tuvieron que resolver a mano durante varios meses los equipos que secuenciaron la ribonucleasa o la insulina, con la diferencia de que sus programas fueron capaces de llegar a la solución correcta en unos pocos minutos para la ribonucleasa.
A lo largo de sus investigaciones recibió fondos de NIH (National Institutes of Health), NSF (National Science Foundation), NASA (National Aeronautics and Space Administration) y la corporación IBM. Estas investigaciones se movían en varios frentes, de los cuáles el primero fue la escritura de una serie de programas en FORTRAN para determinar la secuencias de aminoácidos de las moléculas de proteínas. Estos programas tomaban fragmentos de péptidos obtenidos de la digestión parcial de una proteína, y calculaban todas las posibles secuencias compatibles con los ensamblajes de esos péptidos. Estos programas servían para resolver el mismo puzzle que tuvieron que resolver a mano durante varios meses los equipos que secuenciaron la ribonucleasa o la insulina, con la diferencia de que sus programas fueron capaces de llegar a la solución correcta en unos pocos minutos para la ribonucleasa.
Margaret Dayhoff no fue la única en su época dedicada a la creación de estos primeros programas de ensamblaje. Todos los investigadores que trabajaban en aquella época en este campo se dieron inmediatamente cuenta de que la misma metodología se podrían aplicar al ensamblaje de secuencias genómicas cuando las técnicas experimentales estuvieran disponibles.
Los programas de análisis de secuencias en aquella época siguieron los principios introducidos por el analizador automático de aminoácidos de Stein y Moore. Tanto los programas como el aparato estaban enfocados en recopilar rápidamente una biblioteca de secuencias que pudiera ser usada en estudios de bioquímica comparativa y evolución molecular. Y para promocionar este objetivo, Dayhoff fundó el Atlas of Protein Sequence and Structure (atlas de secuencia y estructura de proteínas), una publicación anual que intentaba catalogar todas las secuencias de aminoácidos conocidas. De forma muy rudimentaria, ésta fue la primera base de datos de información sobre biología molecular, y se convirtió en el recurso indispensable para las primeras investigaciones computacionales.
Con el paso del tiempo (y la creación de internet y la web) esta publicación evolucionó hasta convertirse en 1983 en una de las principales bases de datos online, PIR (the Protein Information Resource). Se convirtió en un importante punto de partida, y un referente para la creación de otras bases de datos basadas en información molecular.
Fuente Madridasd