Una vez que quedó demostrado que las proteínas eran secuencias de aminoácidos donde importaba el orden de los mismos, y que se empezó a secuenciar cada vez más proteínas, el repositorio de información asociado a las proteínas secuenciadas empezó a crecer poco a poco, pero todavía no se habían establecido las correspondencias entre el código genético (el ADN, compuesto de nucleótidos) y las proteínas (compuestas de aminoácidos), ni entre la secuencia de una proteína y su estructura y funcionalidad.
 A finales de 1950 los experimentos realizados por Christian Anfinsen y sus colaboradores mostraron que, una vez desnaturalizada la ribonucleasa (vamos, que perdió su conformación tridimensional y se quedó hecha un hilo), volvía a recuperar de forma espontánea su conformación tridimensional. Con ello quedó asentado el concepto de que la estructura tridimensional de una proteína está determinada única y exclusivamente por su secuencia de aminoácidos.
A finales de 1950 los experimentos realizados por Christian Anfinsen y sus colaboradores mostraron que, una vez desnaturalizada la ribonucleasa (vamos, que perdió su conformación tridimensional y se quedó hecha un hilo), volvía a recuperar de forma espontánea su conformación tridimensional. Con ello quedó asentado el concepto de que la estructura tridimensional de una proteína está determinada única y exclusivamente por su secuencia de aminoácidos.
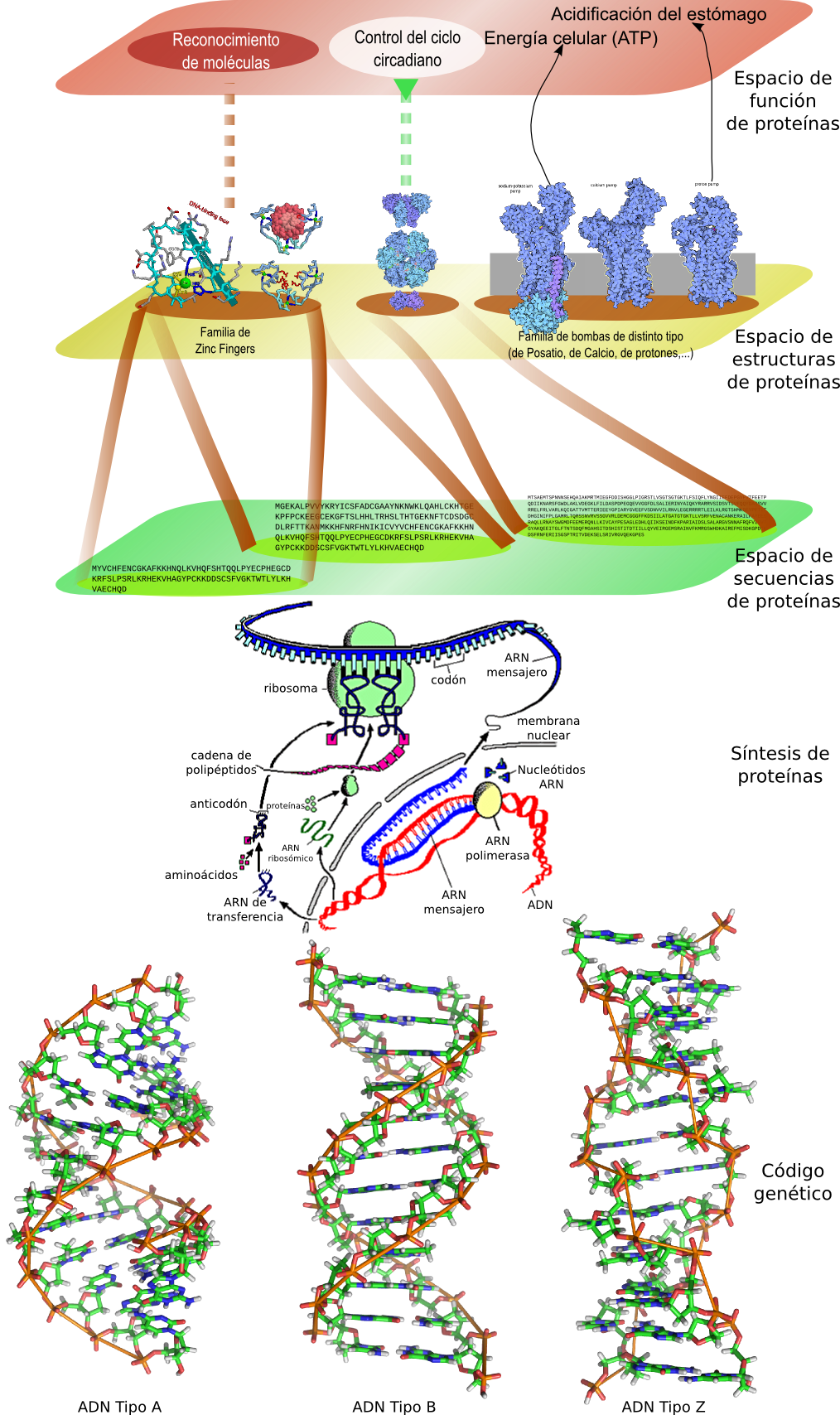
El problema bioinformático que hasta día de hoy no está resuelto de forma general es el de cómo calcular esa estructura tridimensional de la proteína a partir de la secuencia de aminoácidos. Dado que a nivel experimental es muchísimo más fácil secuenciar una proteína que obtener su estructura tridimensional mediante cristalografía de rayos X o resonancia magnética nuclear, la resolución del problema a nivel bioinformático simplificaría mucho la tarea en otras áreas. La estructura de una proteína determina su funcionalidad y participación en el metabolismo de los organismos, y proteínas con estructuras similares juegan papeles similares. Aunque dos proteínas con secuencias muy parecidas tienen siempre estructuras muy parecidas (y por tanto, funcionalidades muy parecidas), dos proteínas con secuencias distintas también pueden tener estructuras similares (por convergencia evolutiva) y por ello funcionalidades parecidas.
Y en la próxima entrega, la aparición de la biología computacional.
Fuente Madridasd
No hay comentarios.:
Publicar un comentario